SSRgenotyper: 简单重复序列的基因分型新工具
SSRgenotyper是一个新开发的免费生物信息学工具,它允许研究人员使用简单重复序列(SSRs)对测序群体进行数字化基因分型,这项任务以前需要耗时的实验室方法。
在最近一期的《植物科学应用》杂志上报道,该工具的开发者设计的程序与目前用于检测和分析SSRs的其他应用程序无缝集成。
简单序列重复是容易发生突变的重复核苷酸短链。这些DNA序列的变异性使其成为基因分析区分个体的理想选择,通常是亲子鉴定和法医检测的首选标记。
在研究领域,SSRS还有一个额外的好处,即它们是选择性中性的,这意味着它们不编码任何物理特征,因此不受大多数类型的自然选择的影响,使它们成为研究种群的绝佳工具,而不会受到趋同进化的模糊影响。
新一代测序的最新进展有助于简化SSR鉴定的过程,特别是在模型生物或具有可用参考基因组组合的群体中。随着技术的不断改进和测序成本的降低,即使在非模式生物中,用于SSR分析目的的大部分基因组测序也变得更加可行,并且在科学文献中得到了广泛应用。
然而,基因分型的过程 — — 确定哪些个体具有哪些等位基因 — — 仍然主要依赖于在电泳凝胶上对扩增的DNA进行可视化,这是一个复杂且具有潜在危险性的过程,因为DNA片段经常被致癌化学物质染色。
它还增加了基于所得条带大小来测量等位基因的问题,这是对扩增的DNA片段中核苷酸数目的估计。由于围绕感兴趣的SSR的侧翼区域可能会有细微的变化,并且由于没有使用这些方法确定等位基因大小的标准化方法,因此无法轻松转移一个实验的基因分型结果或将其与另一实验的基因分型结果进行比较。
该程序不仅减少了对种群进行基因分型所需的时间和工作量,还通过直接计算给定序列重复中的碱基对总数,解决了电泳估计中固有的可转移性问题。
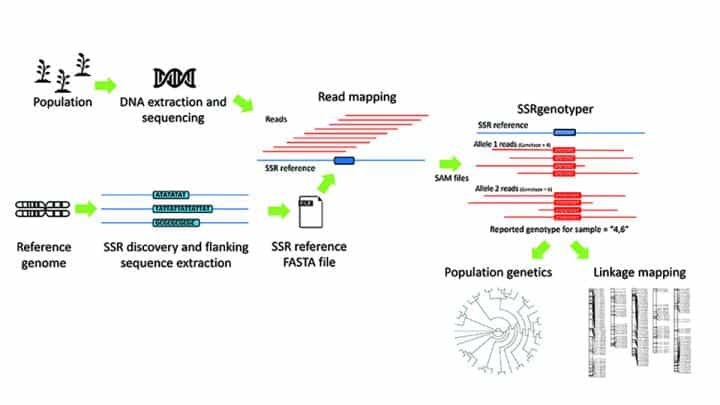
该程序用Python 3编码,运行时只需要三个位置参数,提供了指定几个条件参数的选项(如杂合度的百分比阈值、侧翼区域的大小以及去除虚假等位基因),可以在普通的台式电脑上执行。
一旦完成,SSRgenotyper会生成多种文件类型,包括基本的摘要和统计文件,以及.pop、.map文件以及格式化文件,格式化的文件可用于其他程序,以方便下游分析。
作为概念验证,Maughan和他的同事通过在公开的藜麦(Chenopodium quinoa)和燕麦物种Avena atlantica的序列上运行该程序,测试了SSRgenotyper在正确确定个体基因型方面的准确性。结果准确率为97%或更高,随着加入更多的序列读数,准确率有所提高。获取更多前沿科技 信息 请持续关注:https://byteclicks.com
随着新一代测序方法的不断发展和效率的提高,像SSRgenotyper这样的工具似乎已经准备好减少遗传研究中所需要的实验室工作量。
测序已经是大多数基因研究项目的首选方法,随着成本的不断下降和新的生物信息学工具的开发,未来的群体遗传学研究极有可能完全基于下一代测序工具–完全避免了PCR和电泳的繁琐工作。
SSRgenotyper为全基因组重测序数据提供了一个易于使用,准确且可扩展的SSR基因分型平台。SSRgenotyper可从https://github.com/dlewis27/SSRgenotyper免费获得。


